
プレリリース2023.2.27
科学教育だけでなく、分子デザインのツールとしても活用可能
上智大学(東京都千代田区、学長:曄道佳明)理工学部物質生命理工学科の近藤次郎准教授は株式会社StudioMIDASの中村昇太氏と共同で、科学教育用のオリジナル分子模型「BasePairPuzzle」(図1)を開発し、これを用いた教育プログラムを発表しました。今回発表した分子模型のデータは無料公開されていますので、3Dプリンタさえあれば世界中のどこでも出力して授業などに活用できます。本研究成果は、2023年1月26日に国際学術誌「Journal of Chemical Education」にオンライン掲載されました。
BasePairPuzzleの最も大きな特徴は、ネオジム磁石が組み込んである点です。磁石の引力と反発力が、分子同士が引き合ったり反発したりする力を表現しています。磁石の引力や反発力を自分の手で感じることで、水素結合や静電相互作用の概念を、実感をともなって理解できます。また、2つ以上の分子パーツをくっつけたり離したりして遊ぶことで、DNAがなぜ相補的なアデニン(A)-チミン(T)とグアニン(G)-シトシン(C)という決まった塩基対を形成して遺伝情報を記憶するのかなど、分子レベルのさまざまな生命現象についての理解を深めることができます。
BasePairPuzzleの3Dプリント出力用データ(.stlファイル)および詳しい作り方は下記URLで公開しています。
https://jkondo.wixsite.com/basepairpuzzle/diy
本プレスリリースの末尾には、BasePairPuzzleの実践的な活用方法を記載していますので、ぜひご活用ください。

図1. 3D出力後、彩色したBasePairPuzzle。磁石の引き合う力・反発する感触で、分子同士の相互作用が体感できます。
1953年にジェームズ・ワトソンとフランシス・クリックが報告したDNAの二重らせん構造は一般に広く知られており、構造を学ぶための分子模型キットも数多く市販されています。市販されているDNAの分子模型は、①ゲノムDNAは2本のポリヌクレオチド鎖からできていること、②ポリヌクレオチド鎖は互いに反対方向に伸びていて、DNA全体は右巻き二重らせん構造であること、③2本のポリヌクレオチド鎖はAとT、GとCがそれぞれ水素結合して相補的な塩基対を作ることでつながっていること——の3点を理解することを目的としています。しかし、これまでの研究から、ワトソンとクリックが報告した二重らせん構造は、実はDNAがとりうる構造の一つに過ぎず、生体内でDNAはさまざまな構造をとりうるということがわかっています。
ワトソンとクリックが提唱した二重らせん構造はB型と呼ばれる右向きのらせん構造です。B型以外にも、二本鎖RNAの標準構造として知られるA型、GCGCGC…の連続配列によって形成する左巻きのZ型、さらには、ポリヌクレオチド鎖は通常であれば反対方向に伸びていますが、同じ方向に伸びているものなど、さまざまな種類が報告されています。さらに、三重鎖、四重鎖、八重鎖のような、より複雑な構造も報告されています。こうした構造は、遺伝子発現の制御や病気の発症に関わることから、創薬への応用が期待されています。
このようなDNAやRNAの構造の多様性の背景には、A-TとG-Cの相補的塩基対以外にも、実際には多様な非相補的塩基対を形成できることがあります。さまざまなRNA分子の結晶構造を解析した論文(Leontis et al., 2002, Nucleic Acids Res.)では、約150種類の非相補的塩基対が報告されています。
本研究では、核酸の複雑な立体構造と高度な機能を支える塩基対の多様性と、その背景にある原理を理解することを目的として、3Dプリンタによる分子模型を開発しました。A、T、G、CにRNAでTの代わりに使われるウラシル(U)を加えた5種類の核酸塩基の模型にネオジム磁石を埋め込み、その引き合う力と反発する力を体感することで、塩基間でどのように水素結合が形成されるかなど、身をもって分子同士の相互作用を学ぶことができます。
塩基対は、2つの塩基間の複数の水素結合の距離と角度が、特定の条件を満たしたときにのみ形成されます。水素結合とは、電気陰性度の高い窒素原子や酸素原子と共有結合している水素原子(水素結合ドナー)と、電気陰性度の高い窒素原子や酸素原子の非共有電子対(共有に使われていない電子対:水素結合アクセプター)の間の引力のことを指します。水素結合は静電気力の一種であることから、その強さはドナーとアクセプターの距離や角度に依存しますが、そのことを化学構造式だけから理解することは容易ではありません。そこで、ネオジム磁石のS極とN極をそれぞれ水素結合ドナーと水素結合アクセプターの位置に配置することで、水素結合を磁力として体感できる核酸塩基の分子模型を設計しました。
BasePairPuzzleは、A、T、G、C、Uの5種類のパーツで構成されています。構造の骨格となる炭素原子をAはオレンジ、Cは緑、Gはシアン、UとTは紫、水素結合に関与する水素原子、窒素原子、酸素原子をそれぞれ白、青、赤のボールで表現しています。また、水素結合に関与しない炭素に結合した水素原子は白い棒で表しています。塩基とデオキシリボースやリボースとの結合(グリコシド結合)は各パーツの色の棒で表現しています。各塩基の模型は、3Dプリンタで製作した複数のパーツを組み立てて作ります。ネオジム磁石のS極とN極は、それぞれ水素原子ドナーと非共有電子対の位置に埋め込みます。
BasePairPuzzleの開発では、手のひらに収まるフィット感、磁石がカチッとハマった時の心地よい感覚、そして頑丈さにこだわり、工夫を重ねました。組み合わせたりパーツごとに分解したり試行錯誤することで、分子間相互作用の原理と多様性を学習することができるでしょう。
BasePairPuzzleを教室で使う場合、塩基や原子の種類、水素結合の概念などについては何も説明せず、まずは分子模型に触れさせることをお勧めします。遊んでいるうちに、磁石の力で模型同士が勝手にくっつくようになります。これによって、模型の間(すなわち分子の間)に引きあう力と反発する力があることに自分で気づくのです。そして、模型のどの部分が結合したり反発したりするのかを試行錯誤してみることでしょう。
自由に触る時間を設けた後、何か気づいたことがないか聞いてみると、おそらく「白い玉は赤や青の玉にくっつく」、「白い玉を赤と青の玉の間に入れると、模型同士がくっつく」などの回答が出てくると思います。生徒が自分でこの現象に気づいた後で、水素結合の概念を詳しく説明すると、水素結合が距離や角度に依存する静電的な相互作用の一種であることを身をもって理解できます。
分子模型が磁力によって互いに引き合うことから、4つの塩基はどのような組み合わせでも何らかの形でくっつきます。生徒たちは多様な組み合わせの塩基対を作ることでしょう。2つの塩基の組み合わせだけでなく、3つや4つの塩基を使う生徒も出てくると思います。重要なことは、これらの組み合わせや配置は、私たちの体の中で働いているさまざまなRNA分子中に実在しうる、つまり、すべて正解であるということです。実際にRNAはこれら非相補的な塩基対を形成することによって複雑な立体構造をとり、さまざまな働きを持つことが知られています。BasePairPuzzleを使って、何種類の塩基対が作れるか試してみましょう。
ワトソンは、ダンボールを切って作った4種類の塩基を使って、A-T、G-Cの塩基対が同じ形になることに気づき、DNAの二重らせん構造を発見しました(クリック、ウィルキンスとともに1962年にノーベル生理学・医学賞を受賞)。幾何学的に同一のA-TとG-Cの塩基対を見つけるという課題を与えることで、DNA二重らせんの歴史的発見を生徒に追体験させることができます(図2)。
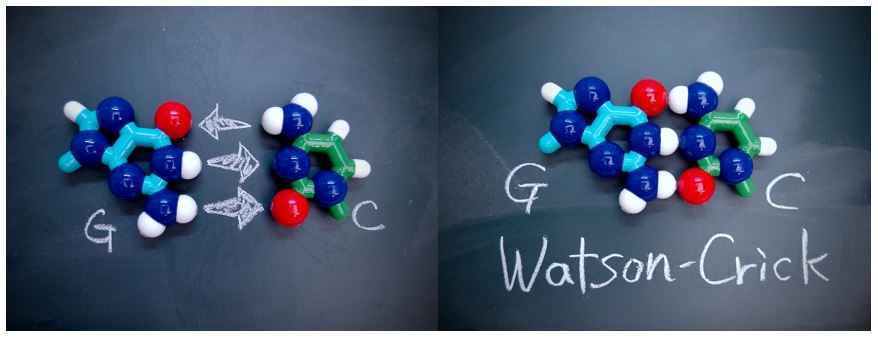
図2. ワトソンが発見したGとCの塩基対。水素結合のドナーとアクセプターが直線的に配置すると水素結合が形成される。
上智大学理工学部物質生命理工学科
准教授 近藤 次郎 (E-mail:j.kondo@sophia.ac.jp)